深層学習によりタンパク質周辺の水和解析を実用速度に
- 創薬への応用を目指し、従来法の計算時間を大幅に短縮 -
2026/02/12
研究室ウェブサイト
発表のポイント
- 創薬において重要なタンパク質周囲の水分子の自由エネルギー分布を高速に計算する、深層学習を用いた新しい解析手法“Deep GIST”を開発しました。
- 薬剤候補のリガンド(注1)結合部位における水分子の自由エネルギー変化の計算において、従来法と良好な相関を示し、水分子の挙動を高速かつ実用的な精度で予測できることを確認しました。
- 創薬において重要な指標である「水分子の置換自由エネルギー」の効率的な評価手法として、薬剤設計への応用が期待されます。
概要
タンパク質は水中でのみ正しい立体構造を保ち、生命現象に不可欠な機能を発現します。タンパク質の結合部位における水分子の振る舞いを理解することは、創薬研究において重要な役割を果たします。従来法のGIST(注2)やWaterMap法は強力な解析手法ですが、計算コストが大きいという課題がありました。
東北大学大学院工学研究科の福島悠朔大学院生(研究当時)と吉留崇准教授は、深層学習を用いることで、これまでGISTで数時間〜数十時間を要していた計算を数十秒で実行できるDeep GISTを開発しました。Deep GISTを用いて、リガンド結合部位の水分子をタンパク質外に移動させるのに必要な自由エネルギー変化を計算したところ、従来法と良好な相関を示し、水分子の挙動を高速かつ実用的な精度で予測できることが確認されました。本手法により、創薬において重要な指標である「水分子の置換自由エネルギー」を効率的に評価できるようになり、薬剤候補の設計や最適化への貢献が期待されます。
本成果は2026年1月23日付けでJournal of Chemical Information and Modelingにオンライン掲載されました。
研究の背景
私たちの体内に存在するタンパク質は、水中でのみ正しい構造に折り畳まれ、生命現象に不可欠な機能を発現します。現在使用されている医薬品の中には、そのメカニズムを利用して、体内の特定のタンパク質に結合することで効果を発揮するものが多くあります。そのため、タンパク質と水分子がどのように相互作用しているのかを解明することは、生命の作動原理を理解する基礎研究のみならず、創薬研究においても極めて重要です。
計算機上でタンパク質と水分子の相互作用を解析する方法の一つに、GISTがあります。GISTを用いると、タンパク質周囲における水の自由エネルギー(水和自由エネルギー)、エネルギー(水和エネルギー)、エントロピー(水和エントロピー)(注3)といった物理量の空間分布を算出できます。これらの分布情報から、特定の領域における水分子の安定性を評価することが可能です。
このような水和自由エネルギーに基づく解析の考え方は、基礎研究にとどまらず、創薬分野にも応用されています。GISTに代表される水和解析の考え方をもとに、創薬の現場で広く利用しやすい形に実装された解析手法であるWaterMap法は、GISTと同様の理論に基づいた解析手法です。WaterMap法は、タンパク質の結合部位などに存在する水分子に着目し、その水和自由エネルギーや水和エネルギー、水和エントロピーの詳細な内訳を評価することで、薬剤設計における重要な指針を提供しています。
しかし、これらの手法には共通して、計算コストが大きいという課題があります。GISTに基づく解析では、タンパク質1構造あたりの計算に数時間から数十時間を要するため、構造が時間とともに変化する「タンパク質の構造ゆらぎ」を考慮した解析への適用は困難でした。また、多数の変異体や類似タンパク質を一括して解析する場合にも、この計算時間の長さが大きな制約となっていました。
今回の取り組み
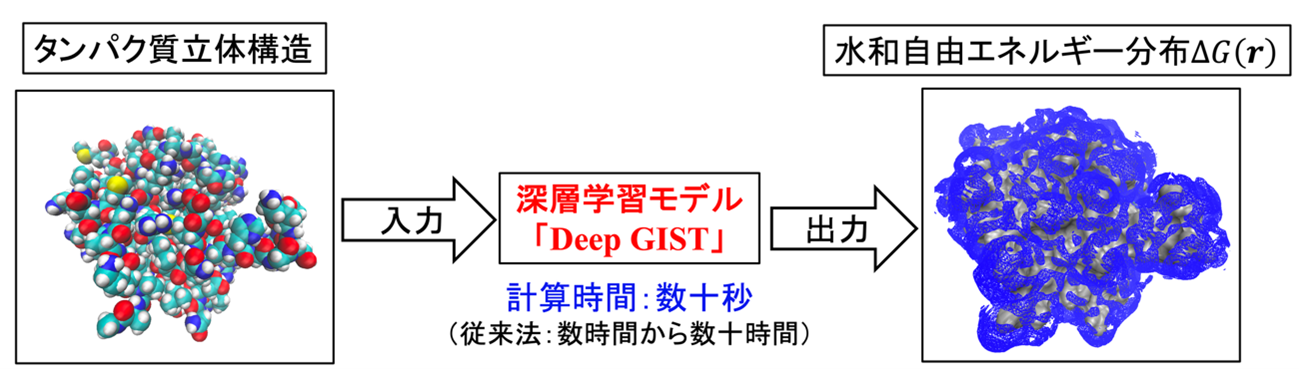
この課題に対し、本研究では深層学習を用いてGIST計算を大幅に高速化する手法の開発に取り組みました。研究グループはこれまでに、タンパク質周囲の水和分布を高速に予測する深層学習モデル“gr Predictor”(注4)を開発してきました。本研究ではこの技術を応用し、タンパク質の立体構造を入力するだけで、その周囲の水和自由エネルギー分布を予測できる深層学習モデルDeep GISTを開発しました(図1)。従来のGIST法では数時間から数十時間を要していた計算が、Deep GISTでは数十秒で完了します。
この手法の有効性を検証するため、Deep GISTを用いて、アシネトバクター・バウマニ由来のペプチジルtRNA加水分解酵素(注5)周囲の水和自由エネルギー分布を予測しました。その結果、従来法(GIST)で特定されていた「水分子が安定に存在できる領域(水和自由エネルギーが低い領域)」を的確に再現できることが確認されました(図2(a))。また、予測精度の指標である決定係数(R2)が0.83という高い値を示しました。さらに、本解析において、Deep GISTによる計算は約29秒で完了し、一方でGISTによる計算は約55時間でした(200 nsの分子動力学シミュレーション:約40時間、タンパク質全体でのGIST計算:約15時間)。これらの結果は、Deep GISTが従来法の計算結果と高い整合性を示しつつ、実用的な短時間で予測を行える手法であることを示しています。
また、Deep GISTの創薬への応用可能性を検証するため、薬剤候補(リガンド)がタンパク質の結合部位に結合する際に、その周囲に存在する水分子がどのように振る舞うかに着目した解析を行いました。具体的には、結合部位に存在する水分子が結合後も保持されるのか、あるいは排除されるのかを予測する計算を行いました。9種類のタンパク質—リガンド複合体を対象に、リガンド結合部位の水分子をタンパク質の外へ移動させるのに必要な自由エネルギーを評価したところ、個々の水分子の自由エネルギーについても、Deep GISTの予測結果は従来法(GIST)の結果と相関係数0.78という高い相関を示しました。さらに、FK506結合タンパク質を用いた具体的な解析では(図2(b))、自由エネルギーが低い(=安定な)水分子は、実際の結晶構造中で観測される水分子の位置と重なり、リガンド結合後も安定して保持されることが確認されました。一方で、自由エネルギーが高い(=不安定な)水分子はリガンドと重なる位置に存在し、リガンド結合に伴い排除される水分子であることを的確に判定できました。これらの結果は、Deep GISTを用いることで、創薬において重要な結合部位周辺の水分子の挙動を、従来法より圧倒的に高速かつ実用的な精度で予測できることを示しています。
今後の展開
Deep GISTは、従来のGIST法や創薬分野で広く用いられているWaterMap法と比べて、短時間で水和自由エネルギー分布を予測できる点に大きな特長があります。この高速性により、従来法では計算コストの制約から困難であった、タンパク質の構造ゆらぎを実際に反映した解析が可能になります。多数の構造スナップショット(時間とともに変化するタンパク質のさまざまな立体構造)に対する一括解析が現実的になり、生体内環境により近い動的な水和状態の理解が進むことが期待されます。
今後、Deep GISTを創薬プロセスに組み込むことで、リガンド結合部位周辺における水分子の挙動を効率よく評価できるようになり、薬剤候補の設計や最適化をより合理的に進められる可能性があります。これにより、従来は試行錯誤に依存していた薬剤設計の一部を、計算に基づいて体系的に検討できるようになることが期待されます。
一方で、GIST法などに基づいて算出される水和自由エネルギーの内訳である水和エネルギーや水和エントロピーについては、個別の予測精度に改善の余地が残されています。今後は、深層学習モデルのさらなる改良や学習データの最適化を進めることで、これらの物理量の予測精度向上に取り組み、より詳細な熱力学解析に基づいた次世代の分子設計技術の確立を目指します。
なお、本研究で開発したDeep GISTのプログラムコードは、研究コミュニティでの活用を促進するため、GitHub(コード共有サービス)にて公開しています。
https://github.com/YoshidomeGroup-Hydration/Deep-GIST
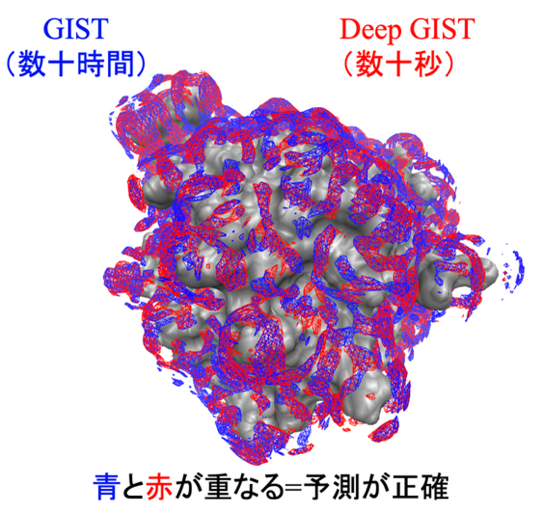
図2. ペプチジルtRNA加水分解酵素周囲の水和自由エネルギー分布を、従来法(GIST、青)とDeep GIST(赤)で計算した結果。従来法の計算時間は、タンパク質全周囲の解析に要する時間であり、200 nsの分子動力学シミュレーションの計算時間を含む。
謝辞
本研究は、科研費「『水和』創薬理論の創生(課題番号:JP21K06107)」、科研費「タンパク質水和の『深層学習モデル』の展開:水和熱力学量の高速計算(課題番号:JP23H04491)」、科研費「『高速・高精度な水和理論』を用いたリガンド結合予測の精度向上(課題番号:JP24K09389)」、科研費「『深層学習に基づく水和理論』を用いたタンパク質シミュレーションの高速化(課題番号:JP25H01505)」の支援により実施されました。また、本研究の一部は、HPCI システム利用研究課題(課題番号:hp210081、hp230022)を通じて、九州大学が提供するスーパーコンピュータシステムITOの計算資源の提供を受け、実施しました。加えて、本研究成果の一部は、大阪大学D3センターのSQUIDを利用して得られたものです。さらに、「東北大学 2025年度オープンアクセス推進のためのAPC支援事業」の支援を受けました。
用語説明
(注1)リガンド
タンパク質に結合する分子で、創薬では薬になる可能性のある分子(薬剤候補)がこれにあたる。リガンドがタンパク質に結合することで、その働きを弱めたり強めたりすることができる。
(注2)GIST
Grid-Based Inhomogeneous Solvation Theory。タンパク質の周囲に存在する水分子の性質を、空間的な分布として解析する計算手法。タンパク質の周りを細かく区切り、水分子が安定に存在しやすい場所や、結合に影響を与える水分子の位置を調べることができ、タンパク質の基礎研究や創薬研究などで活用されている。
(注3)水和自由エネルギー、水和エネルギー、水和エントロピー
本研究では、水和自由エネルギーはタンパク質周囲における水の安定性を示す指標であり、水和エネルギーは水分子同士およびタンパク質との相互作用の強さを、水和エントロピーは水分子の配置や運動の自由度をそれぞれ表す。
(注4)gr Predictor
タンパク質周囲の水和分布を高速に予測する深層学習モデル。プログラムコードは、以下のGitHubにて公開。
https://github.com/YoshidomeGroup-Hydration/gr-predictor
(注5)アシネトバクター・バウマニ由来のペプチジルtRNA加水分解酵素
細菌の一種であるアシネトバクター・バウマニがもつタンパク質の一つ。本研究では、解析手法の性能を検証するためのモデルタンパク質として用いた。
論文情報
著者:福島 悠朔 東北大学大学院工学研究科 応用物理学専攻 大学院生(研究当時)
吉留 崇* 東北大学大学院工学研究科 応用物理学専攻 准教授
*責任著者
掲載誌:Journal of Chemical Information and Modeling
DOI:10.1021/acs.jcim.5c02389